Sabe-se agora que esta era apenas um passo inicial em um longo percurso: para além das letras químicas, a informação também é codificado no modo em que o ADN é empacotado no interior do núcleo da célula. Uma equipa de investigação liderada por Ana Pombo do Max Delbrück Center em Berlim-Buch, em colaboração com colegas internacionais a partir de Itália, Canadá e Grã-Bretanha, já gerou abrangentes mapas 3D da organização espacial do genoma do rato, a partir de células-tronco embrionárias para totalmente desenvolvido neurônios. O trabalho foi publicado na revista Molecular Systems Biology. No futuro, esses mapas podem ajudar a rastrear genes que estão envolvidos em doenças hereditárias.
Diferentes células do nosso corpo carregam a mesma informação genética, escrito na sequência das letras genéticas no nosso DNA. Como os genes dispostos sobre o segmento de ADN são controladas longa tem sido o objecto de muitos anos de investigação. Que os genes são usados numa célula determina se uma célula se desenvolve, por exemplo, na pele, ou uma célula de coração ou do nervo; erros na regulação de genes pode resultar em doença.
Tornou-se claro que o texto linear genética por si só não é suficiente para compreender o genoma. "A organização tridimensional do DNA também é muito importante", diz Ana Pombo, chefe do grupo de pesquisa epigenética regulamento e Chromatin Arquitectura da MDC. Em ratinhos, o segmento do genoma é dividido em 20 pares de cromossomas, que são por sua vez apertada no núcleo da célula de uma maneira não aleatória. "A dobragem espacial complexo do ADN dos cromossomas controla a actividade de genes", explica o cientista.
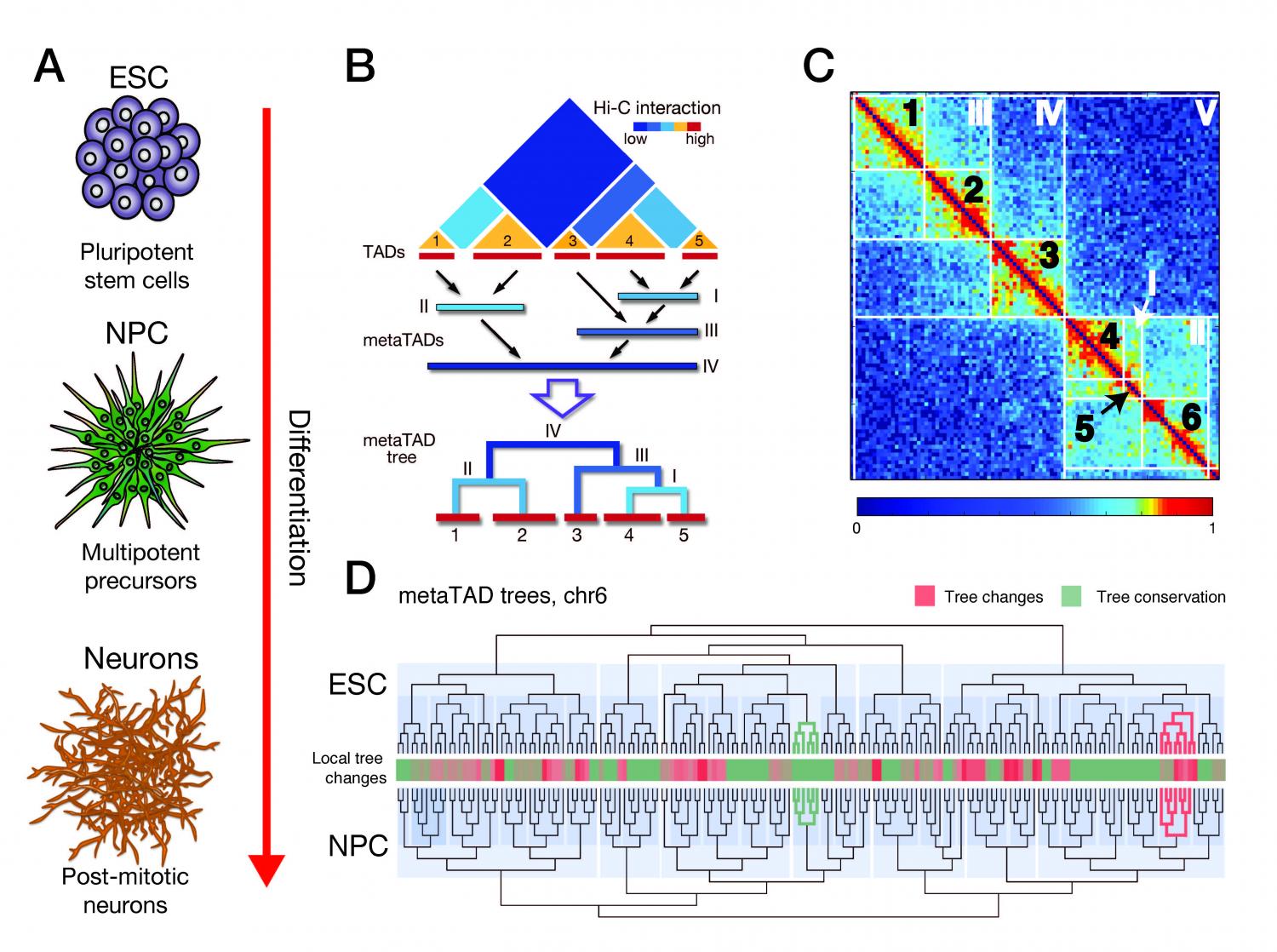
Durante a última década, o progresso tem sido feito considerável na determinação da arquitectura tridimensional de cromossomas. Sabe-se agora que eles são divididos em domínios "topológicos", ou seja em seções de DNA que entram em contato direto com mais freqüência do que os seus vizinhos genômicas. "Até agora a estrutura espacial foi estudada e em torno destes domínios", diz Markus Schüler, pesquisador de pós-doutorado no grupo de Ana Pombo no MDC e um dos primeiros autores do estudo. "No entanto, temos que faltava um quadro completo de como esses domínios interagem uns com os outros e se essas interações estão ligadas a função do gene."
Esta é precisamente a questão abordada no estudo pelos pesquisadores do MDC. Eles levaram uma visão detalhada de como todo o DNA é dobrado nos cromossomos e quais regiões preferencialmente entre em contato com o outro. Como um modelo, que investigou o desenvolvimento de neurónios de rato a partir de células estaminais embrionárias de uma célula progenitora e neurónios diferenciados. Para estes três tipos de células, os pesquisadores analisaram mapas de interação, chamados de dados Hi-C: dados que mostram que as regiões se tocam dentro de cada cromossomo.
Usando essa estratégia, os pesquisadores foram capazes de montar uma matriz de contatos para cada cromossomo em todos os três tipos de células. Os resultados mostraram que os domínios cromossómicas são agrupados em maiores meta-domínios. A dobragem dessas meta-domínios não é aleatória - um ponto crucial. "Várias regiões em um cromossomo vêm juntos, porque eles têm algo em comum", diz Pombo. "As regiões com propriedades funcionais semelhantes entre em contato com o outro, por exemplo, genes que estão ativos, ou que são regulados pelo mesmo mecanismo."
Para ilustrar isso, Pombo usa um fio para representar a forma de seqüência semelhante de DNA. No seu lado, ela constitui várias alças de modo a que o fio se encontra novamente e de novo na base das alças. "Este é o lugar onde conhecer regiões que têm algo em comum", explica ela. O arranjo looping ilustra uma descoberta muito importante: que fornece um meio pelo qual as regiões que estão muito afastadas no ADN linear pode entrar em contacto uns com os outros. "Pela primeira vez fomos capazes de determinar, contactos específicos de longo alcance entre os domínios em todo cromossomos inteiros", diz Pombo.
Os pesquisadores representado essa interação como uma hierarquia de árvore de domínios que mostra que a região está em contato com whih outros. Quando compararam os diagramas de árvore das células-tronco embrionárias, células progenitoras neurais e neurônios eles viram que durante a diferenciação, muitos dos contatos de longo alcance permanecer no local. No entanto, outras regiões formar novos contatos, novamente com base em características comuns. "Mudanças na atividade do gene correlacionam com mudanças na organização espacial", diz Schüler.
Os cientistas acreditam que, no futuro, este mapa de contatos pode ser usado para ajudar a encontrar as causas de doenças genéticas. Por um lado, pode ser utilizado para localizar os rearranjos de cromossomas, que desempenham um papel em condições tais como cancro. Por outro lado, pode ser possível para identificar genes que são responsáveis por doenças congénitas. Este tem sido um resultado de inúmeros estudos genômicos realizados nos últimos anos que ligaram mutações para várias doenças. No entanto, para muitas destas variantes genéticas, os meios pelos quais causam a doença particular, tem sido pouco clara. Eles podem, por exemplo, alterar interações entre genes em vez dos próprios genes.
"Nossos mapas aumentar o leque de alvos no DNA que pode ser afetada por uma única mutação", diz Pombo. Os resultados do grupo tornam possível procurar as outras regiões do DNA com o qual uma variante particular do gene está em contato. Os pesquisadores de Berlim agora quer levar a cabo essas relações para doenças neurológicas, como o autismo e para doenças ósseas.